Librairies Scientifiques¶
Bash¶
La commande bash parallel permet de prendre une liste d’arguments et de les appliquer en parallele à une ligne de commande:
$ ls *.log | parallel pygrep -e ".*ENTRY.*\\d+-\\d+-\\d+.*" {}
La commande time peut donner une idée de la paralélisabilité d’un programmme
$ time
real 0m5.231s
user 0m0.072s
sys 0m0.088s
La règle approximative est:
real < user: The process is CPU bound and takes advantage of parallel execution on multiple cores/CPUs.
real ≈ user: The process is CPU bound and takes no advantage of parallel exeuction.
real > user: The process is I/O bound. Execution on multiple cores would be of little to no advantage.
Profiling¶
profile¶
import cProfile, pstats, io
pr = cProfile.Profile()
pr.enable()
# ... do something ...
pr.disable()
s = io.StringIO()
sortby = 'cumulative'
ps = pstats.Stats(pr, stream=s).strip_dirs().sort_stats(sortby)
ps.print_stats()
print(s.getvalue())
valgrind¶
En plus de valgrind, on peut utiliser kcachegrind qui permet d’explorer les arbres d’appels:
$ valgrind --tool=callgrind <cmd> # assez lent
$ kcachegrind callgrind.out.<pid>
Compilation¶
Certaines librairies telles que Numba permettent de compiler des parties de codes pour en améliorer les temps de calculs.
Le simple ajout du décorateur @jit sur une fonction permet un gain de vitesse
quasi-comparable à un numpy bien compilé
import time
import numpy as np
from numba import jit
# jit decorator tells Numba to compile this function.
# The argument types will be inferred by Numba when function is called.
@jit
def sum2d(arr):
M, N = arr.shape
result = 0.0
for i in range(M):
for j in range(N):
result += arr[i,j]
return result
a = np.random.rand(100,100)
start = time.time()
for i in range(10000):
if i % 100 == 0:
print(i)
sum2d(a)
# np.sum(a)
end = time.time()
print("{:0.3f} s".format(end - start))
Numba est inclus dans Anaconda.
D’autres librairies comparables existent: Nuitka, Parakeet et Hope par exemple.
Parallélisation¶
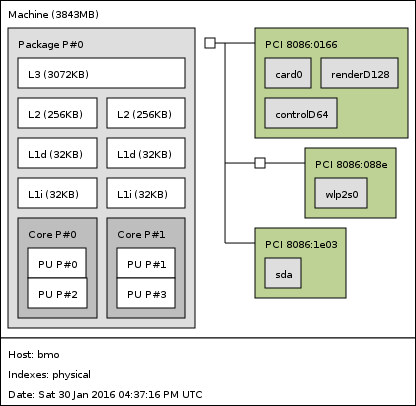
Organisation du processeur¶
Les commandes:
$ hwloc-ls
$ lstopo
permettent de savoir quelle est l’organisation des processeurs et caches. Ça peut donner pas mal d’informations sur la manière de bien optimiser son code.
threading¶
La communication constante entre le programme principal et le thread pour savoir s’il peut switcher vers un autre thread, est la raison pour laquelle le le Threading n’est pas génial en python.
Les sémaphores sont des locks pour lesquels on peut définir un nombre de ressources disponibles.
Les Queues permettente de faire des Piles ou des files d’attentes
multiprocessing¶
En plus des locks, sémaphores et queues, on peut utiliser des barrières et les Pools de processus:
import os
from multiprocessing import Pool
def cube(n):
r = n ** 3
print("(%s) le cube de %s est %s" % (os.getpid(), n, r))
return r
pool = Pool(4)
r = pool.map(cube, range(10))
print(r)
Numpy¶
Il est possible de recompiler Numpy avec des options spécifiques aux architectures matérielles (x86, arm, etc…). On peut, pour certaines opérations gagner un facteur 100 sur les performances.
La commande:
numpy.__config__.show()
permet de voir avec quelle librairies numpy a été compilé.
Les librairies disponibles sont:
ATLAS¶
Prérequis¶
La commande:
$ cat /sys/devices/system/cpu/cpu0/cpufreq/scaling_governor
donne des infos sur la gestion de la fréquence CPU.
Comme on souhaite désactiver la gestion fine de cette fréquence, pour qu’ATLAS ait tout le temps la fréquence maximale, on peut utiliser
$ cpupower frequency-info
$ cpupower frequency-set --governor {performance|powersave}
$ cpupower frequency-set --max <max> --min <min>
$ # Surveillance de la fréquence réelle
$ watch grep \"cpu MHz\" /proc/cpuinfo
Compilation¶
Dans le root d’un virtualenv, créer le dossier packages, dans lequel on place
les sources de ATLAS (décomprésées) et LAPACK (dans son tgz).
$ mkdir <dossier_atlas>/build
$ cd <dossier_atlas>/build
$ ../configure --with-netlib-lapack-tarfile=<path>/lapack-3.6.0.tgz --prefix=<dossier_install> --shared -D c -DPentiumCPS=<freq> -Fa alg '-march=native -Ofast' -Fa acg '-march=native -O3' --force-tids="2 0 1"
$ make
$ # Vérifications des la librairie
$ make check
$ make ptcheck
$ make time
$ # Installation dans <dossier_install>
$ sudo make install
Les options de --force-tids permettent de sélectionner les threads pour chaque coeurs.
Il ne faut en sélectionner qu’un par coeur et pour ça hwloc-ls permet de savoir quels
threads sont associés à quels cores.
Dans l’exemple, on en utilise 2, les threads 0 et 1.
Installation dans numpy¶
# On dit au compilo où chercher la librairie
export LD_LIBRARY_PATH=<dossier_install>/lib:$LD_LIBRARY_PATH
export LIBRARY_PATH=<dossier_install>/lib:$LIBRARY_PATH
# L'installation de numpy se fait avec la librairie compilée
pip install numpy
Fonctions utiles¶
# Créer une matrice initialisée avec des valeurs suivants une fonction
np.fromfunction(lambda i, j: i + 10 * j, (4,4), dtype=int)
a = array([
[ 0, 10, 20, 30],
[ 1, 11, 21, 31],
[ 2, 12, 22, 32],
[ 3, 13, 23, 33]])
# lecture/ecriture de fichiers
np.save('data.npy', a)
np.savetxt('data.txt', a)
b = np.arange(12)
np.savez('data2.npz', a=a, b=b)
# lecture de tous les types
data = np.load("data.npz")
Création d’un nouveau type:
b = np.array([(False, 10.), (True, 5.5)], dtype="b1,f8")
print(b, b.ndim, b.shape, b.size, b.dtype)
[(False, 10.0) (True, 5.5)] 1 (2,) 2 [('f0', '?'), ('f1', '<f8')]
a = b[0]
print(a, type(a), a['f0'], a['f1'])
# équivalent à print(a, type(a), a[0], a[1])
C’est quand même un peu pas terrible comme ça, du coup on peut créer son propre type:
dt = np.dtype([('time', [('min', int), ('sec', int), ('temp', float)])])
b = np.zeros((3, 3), dtype=dt)
print(b)
# [[((0, 0, 0.0),) ((0, 0, 0.0),) ((0, 0, 0.0),)]
# [((0, 0, 0.0),) ((0, 0, 0.0),) ((0, 0, 0.0),)]
# [((0, 0, 0.0),) ((0, 0, 0.0),) ((0, 0, 0.0),)]]
On peut faire des trucs assez chaud pour faire des moyennes par blocks de matrices du genre:
interp_X = X.reshape([SIZE / N, N, SIZE / N, N]).mean(3).mean(1)
interp_Y = Y.reshape([SIZE / N, N, SIZE / N, N]).mean(3).mean(1)
interp_Z = Z.reshape([SIZE / N, N, SIZE / N, N]).mean(3).mean(1)
# Equivalent de ...
interp_X = np.zeros((SIZE / N, SIZE / N))
interp_Y = np.zeros((SIZE / N, SIZE / N))
interp_Z = np.zeros((SIZE / N, SIZE / N))
for i in np.arange(0, SIZE, N):
for j in np.arange(0, SIZE, N):
interp_X[i/N,j/N] = X[i:i+N,j:j+N].mean()
interp_Y[i/N,j/N] = Y[i:i+N,j:j+N].mean()
interp_Z[i/N,j/N] = Z[i:i+N,j:j+N].mean()
Précision numérique¶
sys.float_info donne des informations sur la précision numérique de la plateforme
>>> 0.1 + 0.1 + 0.1
0.30000000000000004
>>> 0.1 + 0.1 + 0.1 == 0.3
False
>>> 0.1.as_integer_ratio()
(3602879701896397, 36028797018963968)
Bon article ici.
Pour limiter les problèmes dans la gestion de devises par exemple, il faut limiter l’utilisation des divisions (ou contrôler les restes) et faire toutes les opérations en centimes.
Celery¶
Répartir un traitement sur plusieurs microprocesseurs, sur la même machine, ou sur des machines différentes (cluster, grid, cloud, etc.).
Celery permet ce genre de topologies, mais de manière transparente pour l’utilisateur. Il suffit de lancer des Workers qui vont se mettre en relation avec un serveur (par défaut RabbitMQ). Celui-ci leur donnera des tâches à faire.
Il faut commencer par définir les tâches à réaliser.
# cel.py
from celery import Celery
app = Celery("SuperDuperApp")
@app.task
def add(x, y):
return x + y
On va ensuite créer un worker
$ celery worker -A cel --loglevel=info
Et lancer notre première tâche
>>> from cel import add
# Lancement normal
>>> add(2, 2)
4
>>> add.delay(2, 2) # envoi de la tâche au worker
<AsyncResult: 2e4b5dff-539c-42f0-9763-5e8ff9041b4d>
>>> add.delay(2, 2).get() # envoi et récupération du résultat
4
On peut également appeler la tâche avec d’autres méthodes que delay
- delay
Envoie la tâche à réaliser directement à un worker. Méthode raccourcie de
apply_async.- subtask
Ces objets permettent de préparer une tâche avant de l’envoyer au worker. Il faut appeler
delaydessus pour envoyer la tâche au worker:>>> s = add.subtask((2, 2), countdown=5) >>> s.delay() # envoi au worker <AsyncResult: 0fa7769c-66d9-4030-b943-5c19afc872f0>
- group
Permet de grouper plusieurs
subtasken un seul appel:g = group(add.s(i, i) for i in range(10)) result = g().get() # Équivalent de g = [add.delay(i,i) for i in range(10)] result = [r.get() for r in g]
- chain
Quand le résultat d’une opération est un argument d’une seconde opération:
# Passe le résultat du add en premier argument de mul c = chain(add.s(2,2) | mul.s(8))
- chord
Regroupe
chainetgroupen un seul appel:chord((add.s(i,i) for i in range(10)), my_sum.s()) # Équivalent à chain(group(add.s(i,i) for i in range(10)) | my_sum.s())